Parte IV: Separação de isoenzimas por electroforese
A — Perspectiva histórica da metodologia
A análise de isoenzimas por electroforese e a sua aplicação na Genética surgiu da confluência de diversas disciplinas (bioquímicas, genéticas e citológicas). Em 1955 descobriu-se que o amido hidrolisado podia servir como matriz de separação electroforética, para a análise de várias amostras em simultâneo [Smithies 1955], não tardando a ser considerada a técnica preferencial para a análise de variações hereditárias a nível molecular, nomeadamente as das hemoglobinas [Lewontin 1982], desde muito cedo o veículo para avanços significativos no estabelecimento dum nexo lógico entre uma variação fenotípica e o seu carácter genético, expresso ao nível proteico e nas suas consequências metabólicas [Pauling et al. 1949, Ingram 1957].
O conceito de isoenzima (também referido como isozima) surgiu pela mesma altura para
referenciar em termos gerais qualquer ocorrência de pelo menos duas variantes moleculares com a mesma
actividade enzimática num indivíduo [Acquaah 1992]
![]() . Embora sem terem actividade catalítica, as
hemoglobinas serviram como exemplo precoce também deste conceito em diversos vertebrados, pela
diversidade alélica na espécie humana, e a sucessão na ontogenia de isoformas separáveis por
electroforese [Lewontin 1982, Harrison et al. 1988]. Em casos como este em que a expressão é distinta
no tempo, ou localizada em órgãos ou compartimentos celulares distintos, verificável por técnicas de
fraccionamento subcelular [Morré 1971], os genes respectivos não são alélicos, pois derivam de
duplicações ocorridas no passado evolutivo da espécie e assim correspondem a loci cromossómicos
distintos, situados na proximidade uns dos outros no mesmo cromossoma ou em grupos de ligação
diferentes [Gottlieb 1982, Acquaah 1992]. Já os isoenzimas codificados por alelos dum mesmo locus são
em geral coexpressos nas mesmas células, designando-se especificamente por aloenzimas ou alozimas
[Acquaah 1992].
. Embora sem terem actividade catalítica, as
hemoglobinas serviram como exemplo precoce também deste conceito em diversos vertebrados, pela
diversidade alélica na espécie humana, e a sucessão na ontogenia de isoformas separáveis por
electroforese [Lewontin 1982, Harrison et al. 1988]. Em casos como este em que a expressão é distinta
no tempo, ou localizada em órgãos ou compartimentos celulares distintos, verificável por técnicas de
fraccionamento subcelular [Morré 1971], os genes respectivos não são alélicos, pois derivam de
duplicações ocorridas no passado evolutivo da espécie e assim correspondem a loci cromossómicos
distintos, situados na proximidade uns dos outros no mesmo cromossoma ou em grupos de ligação
diferentes [Gottlieb 1982, Acquaah 1992]. Já os isoenzimas codificados por alelos dum mesmo locus são
em geral coexpressos nas mesmas células, designando-se especificamente por aloenzimas ou alozimas
[Acquaah 1992].
Se a técnica de preparação de géis de amido para electroforese estava a atingir a sua maturidade [Kristjansson 1963, Fine & Costello 1963], eram poucas as entidades moleculares disponíveis para análise genética: escasseiam as proteínas que podem ser especificamente demonstradas pela sua cor e ao mesmo tempo são abundantes, como é o caso da hemoglobina, enquanto a coloração de todas as proteínas num gel pode revelar muita variação, nem sempre identificável geneticamente por uma análise mendeliana detalhada, e ainda assim restrita às mais abundantes.
A aplicação das técnicas citológicas de revelação enzimática, a amostras submetidas a electroforese, foi o ponto de viragem na análise genética a nível molecular, por trazer para este campo um grande número de marcadores genéticos [Fine & Costello 1963, Scandalios 1964]. A localização microscópica de actividades enzimáticas in situ baseia-se no aproveitamento da especificidade catalítica de cada uma para destacar a sua presença nas estruturas biológicas [Pearse 1968]. Utilizando substratos adequados (os naturais ou seus análogos), a formação dos respectivos produtos resulta directa ou indirectamente na formação dum precipitado corado (ou fluorescente) na vizinhança da actividade enzimática que se pretende localizar. Por electroforese, seguida da incubação do gel num meio de reacção com um substrato específico, revelam-se as diversas localizações desses isoenzimas pela visualização dos precipitados formados pelo produto da reacção ou seu derivado.
Em 1964, quando foi publicado o primeiro estudo usando a metodologia de separação de isoenzimas com fins de análise genética, o número de actividades enzimáticas para as quais existiam protocolos de revelação histoquímica era ainda relativamente limitado [Davenport 1960, Pearse 1968], mas não deixava de abrir uma perspectiva de aumentar em muito o número de loci disponíveis para análise. Esse primeiro estudo fez a demonstração, em géis de amido após electroforese, de variações na distribuição da actividade de fosfoglucomutase (EC 5.4.2.2) entre amostras de diferentes indivíduos da espécie humana [Spencer et al. 1964]; identificou-as com diferentes alelos e verificou que as frequências genotípicas não se afastavam significativamente da distribuição de Hardy-Weinberg. Quase de imediato seguiram-se estudos análogos abrangendo várias actividades enzimáticas em simultâneo [Hubby & Lewontin 1966, Lewontin & Hubby 1966], tendo-se verificado que, quer em animais, plantas ou microorganismos havia um grande número de loci enzimáticos polimórficos, sendo cada indivíduo heterozigótico numa proporção nada irrisória dos loci que se estudavam [Nei 1987].
Paralelamente, a decifração do código genético [Khorana et al. 1966] e as primeiras sequências de proteínas [Canfield 1963], a par do conceito de colinearidade entre estas e as dos nucleótidos nos cromossomas [Crick 1958], trouxeram uma perspectiva estrutural à variação isoenzimática, a qual se deve essencialmente a substituições de aminoácidos com implicações no ponto isoeléctrico, por sua vez resultando de mutações no material hereditário [Acquaah 1992]. Em consequência, a dinâmica da variação evidenciada na análise de isoenzimas por electroforese passava a entender-se dentro da perspectiva da dinâmica evolutiva das populações, fazendo desta metodologia o centro das atenções da Biologia Evolutiva durante alguns anos [Lewontin 1973, Nei 1987]. Com o progressivo aumento da utilização dos métodos de análise das sequências do DNA é que a análise isoenzimática perdeu esse protagonismo, porém retendo alguns nichos de utilização preferencial [May 1991].
B — Aplicação genética do método
1) Consequências para a Genética de Populações e a Biologia Evolutiva
O frequente polimorfismo nos loci isoenzimáticos observado por análise isoenzimática abriu um campo vasto de aferição dos pressupostos da teoria genética das populações. E se trouxe a possibilidade de estudar cada população com respeito a um número elevado de loci em simultâneo, permitindo através das estatísticas da teoria genética descrever e interpretar aspectos da sua subdivisão, do fluxo de genes, de modelos de reprodução, da existência de selecção, etc., por outro também deu origem a uma importante polémica sobre a interpretação desses polimorfismos — o debate entre “seleccionismo”e “neutralismo” [Roughgarden 1979]. A teoria neutral da variação molecular [Kimura 1983, Nei 1987] lançou esse debate ao reviver e actualizar diversas fontes teóricas, nomeadamente a chamada “escola clássica” de Müller sobre as mutações [Dobzhansky et al. 1977, Kimura 1983, 1991], o modelo de Fisher da tolerância às mutações em função do fitness [Fisher 1958, Kimura 1983] e o chamado “dilema de Haldane” sobre os custos demográficos da substituição de um alelo por outro mais favorável [Dobzhansky et al. 1977, Kimura 1983]. Com base no efeito combinado do aparecimento de novos alelos (por mutação) e da fixação de variantes (por deriva genética), no decurso de tempos evolutivos suficientemente longos e sem a intervenção de processos selectivos, essa teoria explicava a variação na estrutura primária das proteínas entre diferentes táxones, assim como o polimorfismo nos loci isoenzimáticos, postulando que uma grande parte dos alelos fixados numa população resultaram de mutações selectivamente neutrais, enquanto a nível molecular a acção da selecção natural, aparte a depuração de mutantes deletérios [Kimura 1983, 1991], só episodicamente envolve novos alelos favoráveis. Por outras palavras, o valor selectivo da variação a nível molecular que se observa nas populações — ou entre populações dentro duma espécie — é em princípio neutro, não excluindo que haja exemplos de ser mantida por mecanismos selectivos, mas definindo-os como excepções.
Tais propostas vieram pôr em causa muito do arrazoado neodarwinista sobre adaptação [Lewontin 1985]. Independentemente de aceitar-se ou não a chamada teoria neutral da variação a nível molecular, justifica-se a importância que adquiriu na Biologia pelo rigor matemático com que permite analisar os polimorfismos nas mais diversas situações, e formular hipóteses nulas para a realização de testes estatísticos à variação molecular [Kimura 1983, Fuerst et al. 1977; Chakraborty et al. 1978, 1980, Nei 1987]. Contudo, a noção de que a maior parte dos polimorfismos enzimáticos é neutral, a par da conveniência desse pressuposto para a aplicação de estatísticas populacionais donde se pretendem inferir processos evolutivos, acaba por levar à convicção, hoje mais ou menos generalizada, de que a priori os polimorfismos isoenzimáticos são selectivamente neutrais. São contudo várias as fontes potenciais de erro que não são acauteladas nesta atitude.
Tradicionalmente, só se pode considerar que um estudo com polimorfismos enzimáticos fez uma amostragem representativa do genoma quando abarque duas ou mais dezenas de loci, com resultados médios comparáveis a outros estudos da mesma natureza, na mesma ou noutras espécies [Lewontin 1973, Nei 1987, DeWoody & DeWoody 2005]. Subjacente a esta exigência estão diversos factores, entre os quais avulta a heterogeneidade entre diferentes loci, reflexo da maior ou menor influência que a variação em cada um pode ter no fitness dos indivíduos (isto é, do impacto fisiológico dos alelos desfavoráveis), e que se traduz numa variável taxa de substituição por fixação [Nei 1987, Kimura 1991]. Mas essa heterogeneidade pode também implicar a possibilidade do polimorfismo envolver, numa minoria de loci, factores adicionais ao modelo básico mutação-deriva (por exemplo, selecção). Além disso, e aqui residirá a maior limitação prática à Teoria Neutral, especialmente quando se trate de espécies com longos intervalos entre gerações, é a das populações não se encontrarem em situação de equilíbrio entre mutação e deriva (e migração), o que torna inválidas as inferências que assumem esse equilíbrio.
Ajustamentos teóricos no discurso neodarwinista vieram responder a algumas críticas neutralistas [King 1967, Sved et al. 1967, Wills 1978, Johnson 1979], mas a afirmação do papel da selecção natural nos polimorfismos enzimáticos, e noutros tipos de loci, tem envolvido demonstrações nem sempre triviais na prática, se bem que muito instrutivas. No caso da fosfoglucose isomerase, sabe-se que entre os diferentes alelos há importantes variações da eficiência catalítica e da estabilidade termodinâmica, que têm implicações fisiológicas diferentes em diferentes fases do desenvolvimento [Watt et al. 1983, Watt 1992, Liu et al. 1999]; e a persistência de polimorfismos da cadeia β da hemoglobina humana em zonas onde existe malária está associada a maior fitness dos heterozigóticos nessas zonas [Harrison et al. 1988]. Um exemplo muito bem estudado onde se conjugam diversos argumentos de ordem genética, fisiológica e evolutiva para atribuir a vantagem dos heterozigóticos como factor determinante do elevado polimorfismo é o do conjunto de loci, localizados na região MHC do genoma humano, envolvidos na apresentação de antigénios aos receptores dos linfócitos T: o estudo do genoma humano sequenciado identificou esta região como a única onde existe um excesso de variabilidade em relação ao que seria previsível pela Teoria Neutral [International SNP Map Working Group 2001] — um exemplo análogo em plantas é o de loci envolvidos na autoincompatibilidade [Charlesworth 1995, Uyenoyama et al. 2001].
A categorização dos polimorfismos enzimáticos, segundo o tipo de metabolismo onde intervêm em diversas espécies animais, revelou padrões que parecem longe de ser fortuitos [Johnson 1974]: i) as actividades enzimáticas que utilizam eficientemente diversas moléculas como substrato de reacção, isto é, que têm um largo espectro de especificidade (caso de várias hidrolases), ii) os de especificidade estrita mas com função reguladora do metabolismo, seja do fluxo através da respectiva via metabólica, seja do funcionamento de várias vias metabólicas através das concentrações disponíveis de NAD(P)H ou ATP (fosfoglucomutase, fosfoglucose isomerase, glucose 6-fosfato desidrogenase, adenilato quinase, enzima málico, etc.), e iii) os de especificidade estrita e sem função reguladora relevante (definidos cineticamente pelo facto das respectivas reacções se encontrarem próximas do equilíbrio in vivo). A maior parte do polimorfismo encontrava-se nas duas primeiras categorias, tendo sido argumentado por Johnson que isso se explica pela vantagem selectiva de haver formas alélicas que alarguem o espectro de substratos originários do ambiente externo que são metabolizados eficientemente (categoria i), ou que aumentem a eficiência do metabolismo face a variações ambientais (nomeadamente em populações que abrangem clines de temperatura ou doutros factores, categoria ii). A generalizar-se esta categorização, pode não ser possível comparar os resultados de estudos que fizeram a amostragem de diferentes conjuntos de loci enzimáticos, até na mesma espécie; mas não parece que a ideia tenha sido retomada, sequer pelo seu próprio autor (contudo, ver as conclusões de Zanetto et al. [1993], que são analisadas na secção 2, “Alguns exemplos em Quercus”).
Foi ainda Johnson que, a propósito da interpretação de polimorfismos enzimáticos em plantas, apontou a necessidade de avaliar quaisquer modelos de selecção na perspectiva do que designou por fenótipos metabólicos, destacando entre outros o fluxo energético pela glicólise e pela gluconeogénese, a adaptação à anaerobiose nas raízes sujeitas a alagamento, ou o ciclo de Calvin da fotossíntese [Johnson 1979]. Por seu lado, Hamrick [1979] analisou exemplos de plantas onde se notam variações das frequências de certos alelos em áreas relativamente próximas, associados ou não a variáveis ambientais, mas quase sempre com excesso de heterozigóticos em relação aos valores da distribuição de Hardy-Weinberg, o que pode sugerir selecção favorável a esses genótipos.
Apesar de tudo, a análise crítica dos resultados com isoenzimas (e com proteínas não-enzimáticas, analisadas também por electroforese) num grande conjunto de populações animais mostrou persuasivamente a quase suficiência do modelo mutação-deriva como moldura teórica [Fuerst et al. 1977; Chakraborty et al. 1978, 1980]. Adicionalmente, também permitiu afirmar que a variação isoenzimática resulta provavelmente do acumular duma colecção potencialmente infinita de variantes mutacionais independentes (infinite-allele), e não por variações sucessivas (stepwise mutation), mas com diferentes taxas de mutação entre diferentes loci [Chakraborty et al. 1980]. Apesar da força dos argumentos estatísticos, há sempre excepções, nem sempre atribuíveis a erros estocásticos, e permanece sempre a possibilidade das forças selectivas actuando num locus passarem despercebidas por diluição num conjunto de dados predominantemente neutral, o que aliás, pela sua heterogeneidade, pode explicar a incompatibilidade dos modelos de vantagem selectiva dos heterozigóticos, ou da persistência de alelos ligeiramente desfavoráveis (slightly deleterious), com as distribuições de conjunto. No entanto, em nenhum caso se vai além de afirmar que a maior parte dos polimorfismos são neutrais, não que o sejam na totalidade. Por outro lado, mesmo que por hipótese todos o sejam, constituem apenas uma pequena amostragem do genoma e por isso as estimativas de heterozigose que são feitas a partir da sua análise dão valores pouco correlacionados com o valor real [DeWoody & DeWoody 2005].
Assim, em rigor há que assumir a tarefa de demonstrar se, nas populações duma espécie, o polimorfismo num determinado locus se deve a pressões selectivas desfavoráveis à fixação de qualquer dos alelos existentes, seja por vantagem dos heterozigóticos, por selecção dependente das frequências alélicas ou pela chamada selecção diversificante [Dobzhansky et al. 1977, Liu et al. 1999]. É nesse sentido que vai a recomendação de aplicarem-se as abordagens da chamada “genética demográfica”, nomeadamente pelo acompanhamento das frequências alélicas e genotípicas ao longo do ciclo de vida [Clegg et al. 1978, Hamrick 1982, Mitton 1993].
Das várias estatísticas usadas para descrever os polimorfismos nas populações, destacam-se a percentagem de loci polimórficos na população (P) e a probabilidade esperada de loci heterozigóticos em cada indivíduo, que se representa com a média entre os diferentes loci, monomórficos incluídos, das frequências de heterozigóticos esperadas He (isto é, da distribuição de Hardy-Weinberg), e o número efectivo de alelos por locus (Ae, igual ao inverso da frequência de homozigóticos esperada, 1–He). Uma fórmula de He é [Hamrick 1979]
![]() , sendo pij a frequência do alelo j do locus i com ki alelos
, sendo pij a frequência do alelo j do locus i com ki alelos
![]()
Na literatura proveniente da Alemanha é utilizado normalmente um sistema alternativo de parâmetros (e designações próprias) desenvolvido por Gregorius e colaboradores [Hattemer 1991], incluindo a diversidade do fundo genético ν (algebricamente idêntica a Ae para cada locus; entre loci, usa-se a média harmónica, ou então a diversidade gamética hipotética V=Πνi) e a diferenciação populacional total δT (quase idêntica a He para cada locus, usando-se a média aritmética entre loci).
As plantas de semente apresentam a maior diversidade de valores estatísticos, atribuível pelo menos em parte às diferenças entre sistemas reprodutores. Há uma tendência geral de aumento da P com os valores de He (como esperado), em associação com algumas características, em especial a fecundidade e o modo de polinização, com as quais se correlacionaram significativamente outras características da reprodução, designadamente as preferências de cruzamento, tempo de geração e mecanismo de dispersão das sementes, assim como o estádio de sucessão ecológica, expansão geográfica e habitat, e o número diplóide de cromossomas ([Hamrick et al. 1979], tabela 2.5).
Tabela 2.5 — Resumo das características estudadas por Hamrick et al. [1979] em relação às tendências de He em plantas. Considera-se alto ou baixo He quando este, para o estado de carácter em causa, se situa 2 vezes o erro-padrão acima ou abaixo do valor médio global (0,135). Entre os caracteres ausentes desta lista, refira-se o mecanismo de dispersão das sementes (He decrescente de aladas/plumosas, grandes, ingeridas, pequenas, até aderentes aos animais) e o modo de reprodução (sexuada vs. assexuada, sem qualquer correlação com os valores de He).
Tendências |
Alto He (≫ 0,135) |
Baixo He (≪ 0,135) |
Estados de carácter |
Fecundidade alta (> 104/geração) Polinização pelo vento Predominância de cruzamentos ou modo misto (cruzamentos e autocruzamentos) Plantas perenes de geração longa Estádio sucessional tardio 2n > 30 cromossomas |
Fecundidade média-baixa (102–103/geração) Autopolinização Preferência pelo autocruzamento Plantas bienais Endémicas Xéricas |
O género Quercus coincide totalmente com as tendências para alto He, mas a análise isoenzimática não tende a confirmá-lo, pelo menos em comparação com os valores das coníferas [Sork et al. 1993b]. Este facto pode lançar algumas dúvidas sobre a representatividade destes valores face à diversidade genética real dos Quercus, ou pelo menos à que se revela em caracteres fisiológicos através de ensaios de proveniência, implicando uma variabilidade adaptativa intra-específica muito importante [Kriebel 1993, Kleinschmit 1993, Sork et al. 1993b, Almeida et al. 2005].
Coeficientes de fixação, e divergência e fluxo genéticos dentro das populações
Os factores determinantes do polimorfismo selectivamente neutral são em primeiro lugar a mutação e o tamanho efectivo Ne, tais que o valor de equilíbrio do coeficiente de fixação é F = 1/(1+4Neu), [Nei 1987], onde u designa a taxa de mutação para a unidade genética em causa, seja ela locus, codão, nucleótido, etc.. A acção permanente da mutação não permite a fixação absoluta (F = 1), e o mais provável será resultar polimorfismo sem implicar pressão selectiva se o valor de Ne ou o de u (ou de ambos) for bastante elevado — os chamados micro-satélites ou sequências simples repetidas (SSRs) constituem um exemplo de taxa de mutação elevada [Chakraborty et al. 1997], mas os loci enzimáticos não [Nei 1987]. O estudo das distribuições das frequências alélicas e do número de alelos por locus enzimático ajusta-se satisfatoriamente ao modelo neutral (mutação-deriva) [Chakraborty et al. 1980], excepto em casos onde há um excedente de alelos raros (usando para critério de raridade uma frequência inferior a 5%), que se presume representarem situações de não-equilíbrio na sequência de bottlenecks evolutivos.
Pela fixação, um valor limitado de Ne implica divergência em relação a outras populações da mesma espécie, que pode ser mais ou menos atenuada pelo fluxo genético entre elas. O mesmo processo pode até dar-se dentro duma população — ou seja, a subdivisão das populações em unidades panmícticas, entre as quais o fluxo genético é mais ou menos limitado — acarretando uma diversificação interna (estratificação) suficiente para manter polimorfismos. Por outro lado, a heterogeneidade das distribuições subpopulacionais de frequências, gerada pela deriva genética, traduz-se num decréscimo da frequência de heterozigóticos observados ao nível da população (Ho) em relação ao previsto pela distribuição de Hardy-Weinberg (He), o chamado efeito de Wahlund [Wright 1969]. Este decréscimo é expresso em termos de coeficiente de fixação pela fórmula F = 1 – Ho/He [Wright 1969, Berg & Hamrick 1997] e, para um alelo de frequência p no conjunto da população, é igual ao dobro da variância da respectiva frequência p' (na geração anterior) entre as diferentes subpopulações [Wright 1969]: Ho = 2p(1 – p) – 2(σp')2, donde F = (σp')2/[p(1 – p)]; assim, F é igual a 1 quando a variância atinge o seu valor teórico máximo, isto é, quando em todas as subpopulações esse alelo está fixado ou se perdeu.
Como as subpopulações têm Ne limitados, mesmo que funcionalmente sejam panmícticas, têm coeficientes de fixação próprios; neste quadro, Wright propôs que se considere um coeficiente de fixação para a população no seu todo (FIT), e outro para as subpopulações (FIS) [Wright 1969, Nei 1987]; FIS assume-se idêntico entre subpopulações e tenderá a ser maior que 0 porque os valores de Ne nas subpopulações são limitados, mas ainda assim FIT > FIS pelo efeito de Wahlund, e para expressar este efeito de diversificação populacional usa-se a estatística FST = (FIT – FIS)/(1 – FIS). Embora na formulação original se obtivesse uma estimativa de FST para cada alelo (cf. p. ex. Chakraborty et al. [1977]), a reformulação dos cálculos de FIT e FIS em função de frequências observadas e esperadas de heterozigóticos permite obter valores por locus [Nei 1987]. Além disso, a reter-se o conceito mais lato das estatísticas F, não só como coeficientes de fixação mas como medidas de correlação entre gâmetas [Wright 1965], FST representa a priori uma medida da diferenciação genética entre subpopulações que pode ou não ser neutral [Nei 1987]. FST também pode ser calculado segundo uma perspectiva de análise de variância, como a proporção da variância total nas frequências que é atribuível às subdivisões [Weir & Cockerham 1984].
Como o processo de fixação de genes é independente entre diferentes subpopulações, a
transmissão de gâmetas entre elas tende a homogenizá-las, com o aparecimento de heterozigóticos nos
loci onde se acumularam divergências. Wright demonstrou, para o chamado modelo das ilhas (um
número bastante grande de subpopulações de dimensão Ne constante e com uma taxa de migração m
comum a todos os fluxos genéticos por migração [Nei 1997, Sork et al. 1999]), que FST = 1/(1 + 4Nem)
[Wright 1965, Nei 1987, Berg & Hamrick 1997]
![]() . O valor de Nem quantifica por isso o grau de
homogeneização entre as subpopulações em situações de equilíbrio entre migração e deriva e,
complementado por outras estatísticas, permite avaliar a que ponto a migração pode contribuir para o
polimorfismo na população, assumindo-se algo arbitrariamente que é suficiente para tal quando superior
a 1 [Mills & Allendorf 1996]. Mas esta relação entre Nem e FST refere-se apenas ao modelo das ilhas e
assumindo neutralidade selectiva dos polimorfismos, o que não parece obstar a um uso algo
indiscriminado [Sork et al. 1999].
. O valor de Nem quantifica por isso o grau de
homogeneização entre as subpopulações em situações de equilíbrio entre migração e deriva e,
complementado por outras estatísticas, permite avaliar a que ponto a migração pode contribuir para o
polimorfismo na população, assumindo-se algo arbitrariamente que é suficiente para tal quando superior
a 1 [Mills & Allendorf 1996]. Mas esta relação entre Nem e FST refere-se apenas ao modelo das ilhas e
assumindo neutralidade selectiva dos polimorfismos, o que não parece obstar a um uso algo
indiscriminado [Sork et al. 1999].
O coeficiente de diversidade genética GST [Nei, 1973] tem um significado análogo ao FST mas sem depender de coeficientes de fixação nem implicar um modelo “ideal” de subdivisão populacional [Nei 1997]; a heterogeneidade das frequências dos alelos em cada locus, entre subpopulações duas a duas, é convertida nas respectivas distâncias genéticas, donde se deduz, dos heterozigóticos esperados HT (equivalente a He na nomenclatura de Nei), uma componente DST que quantifica a diversificação entre subpopulações (baseada no cálculo dos quadrados das diferenças pij–pik, entre as frequências de cada alelo i entre todos os pares {j,k} de subpopulações). A proporção de HT atribuível a DST é o valor de GST. Uma extensão deste sistema, no sentido de individualizar componentes de diferenciação entre espécies e entre regiões, foi proposto num estudo sobre Quercus petræa e Q. robur [Bodénès et al. 1997].
O sistema de Gregorius adopta uma medida de distância genética d0 baseada no módulo das diferenças pij–pik, e uma diferenciação genética Dj, de cada população j em relação às restantes, que é o valor de d0 quando o índice k se define para o conjunto das restantes [Hattemer 1991]. A média dos Dj (ponderada pelo tamanho de cada subpopulação) dá a diferenciação entre subpopulações δ, cujo significado será análogo a GST e FST, embora geralmente dê valores numericamente superiores [Herzog 1996, Gehle 1999].
Existe ainda uma outra abordagem genética à diferenciação entre populações, que se baseia nas frequências dos alelos só existentes numa região ou mesmo numa só subpopulação (“raros” ou “privados”), cujo desaparecimento nas restantes se postula dever-se à deriva genética não compensada pelo fluxo migratório (evidentemente, para demonstrar-se esta situação são necessárias amostragens de grandes dimensões [Neigel 1997]). A média das frequências destes alelos “raros” p(1) relaciona-se com Nem através da igualdade ln[p(1)] = a·ln(Nem) + b, sendo a e b valores dependentes da dimensão de amostragem [Slatkin 1987, Neigel 1997].
As estatísticas de diferenciação entre subpopulações são formuladas para cada locus e, por via disso, o aproveitamento de toda a informação disponível de vários loci é em geral limitado a assumir-se que toda a variação é neutral, sujeita a um único conjunto de factores (excepto as taxas de substituição), e em condições de equilíbrio, para então adoptar-se a média entre loci. É improvável que isso se verifique em algum caso, e por isso as interpretações associadas a essas médias multi-locus podem estar completamente erradas. Podem adoptar-se várias estratégias, como por exemplo para levar em conta o desequilíbrio gamético na diferenciação entre populações [Kremer et al. 1997], ou simplesmente para análise hierarquizada sobre factores ecológicos ou outros [Gram & Sork 2001] (cf. “Alguns exemplos em Quercus”, mais adiante). O desenvolvimento de novos marcadores e novas abordagens de análise [Vekemans & Hardy 2004, Smouse & Sork 2004], geralmente sem recurso a loci isoenzimáticos, está-se a revelar uma melhor solução (cf. parte II secção D1).
Não menos significativa é a demonstração de que as estimativas de m podem ser fortemente enviesadas se a distribuição de frequências no pólen real for diferente da estimada, ou for diferente da dos gâmetas femininos, ou se Ne for muito inferior ao número de indivíduos da população (o que acontece, nomeadamente, se o contributo dos diferentes indivíduos para a reprodução for variável) [Burczyk & Chybicki 2004].
O pressuposto de neutralidade nos polimorfismos aloenzimáticos
A relação teórica entre Nem e FST, colocada como o paradigma de base para a medição indirecta dos fluxos genéticos nas populações, pelo menos como complemento ou até em substituição de medições directas baseadas por exemplo no cálculo de dispersão de sementes, distâncias percorridas pelos polinizadores, etc. [Slatkin 1987, Sork et al. 1999], é especialmente controversa. Para além da duvidosa relevância das estimativas de Nem, por causa da provável inaplicabilidade dos seus pressupostos nas populações reais (modelo de isolamento pela distância, equilíbrio neutral entre mutação, deriva e migração [Wright 1965, Nei 1997]), é evidente a arbitrariedade das interpretações, quando alguma vez são propostas. Numa discussão do pseudo-parâmetro Nem obtido de FST [Slatkin 1987], cita-se um exemplo com borboletas da espécie Euphydrias editha, onde os valores de FST obtidos variaram entre 0,017 e 0,291 consoante o locus. Se se adoptar a “regra” de que os valores de Nem (calculados a partir de FST) inferiores a 1 indicam um fluxo genético reduzido entre as subpopulações, e face a ter havido apenas um valor de Nem nessas condições (0,6 correspondente ao FST de 0,291), comparado com sete valores Nem iguais ou superiores a 3,8, Slatkin concluiu que só o locus em causa (ou outro muito proximamente ligado, pelo chamado efeito hitch-hike) estaria implicado na adaptação local de cada subpopulação (diferentes alelos nesse locus eram favorecidos em diferentes subpopulações, contrariando o efeito tendencialmente homogeneizador do fluxo genético entre subpopulações). Considerando os valores de Nem nos restantes loci como estimativas aproximadas do “verdadeiro” valor, Slatkin adoptou o valor médio de 7,8 e passou a analisar a aparente contradição com medidas directas feitas na mesma população, que colocariam Nem de maneira convincente num valor da ordem dos 0,1; introduzindo considerandos de ordem geológica, nesta e noutras espécies, tendentes a colocar a divergência entre as subpopulações num passado muito recente, concluiu que era insuficiente para a diferenciação nos sete loci considerados neutrais, mas suficiente para a diferenciação adaptativa no oitavo locus.
Noutro caso de aparente contradição entre as estimativas directas de fluxo genético entre subpopulações e os valores Nem obtidos dos FST em loci enzimáticos e serológicos, respeitante à subdivisão em castas dos Dhangars (Maharashtra, União Indiana) [Chakraborty et al. 1977], os autores chamaram a atenção para o número de gerações desde a instituição do sistema de castas, na ordem de algumas centenas, dando a entender um processo ainda incipiente de diferenciação genética entre castas para justificarem o facto dos valores de FST serem demasiado baixos em relação à situação de equilíbrio entre migração e deriva. Mas o Ne médio de 4000 proposto por casta será discutível, não só pela presumível variação do Ne entre castas, que viola um dos pressupostos para o cálculo de FST, mas também pela duvidosa aplicabilidade desse valor no longo período de tempo em causa; se por hipótese o Ne fosse mais baixo, menos incipiente seria o processo neutral de diferenciação (o número de gerações necessário é proporcional ao Ne [Kimura 1983, Nei 1987]) mas, ao mesmo tempo, mais elevada seria a estimativa de m, tornando a sua contradição com as estimativas directas ainda maior. Também aqui persiste a dúvida sobre a suficiência da explicação neutral para os valores de FST observados.
A questão sobre as actuais populações das regiões temperadas do Hemisfério Norte não se encontrarem em equilíbrio neutral (mutação-deriva), especialmente quando se trata de espécies com tempos de geração longos, parece geralmente plausível face à recorrência de períodos glaciares e consequentes migrações e persistência em refúgios confinados, acarretando reduções drásticas dos Ne (efeito bottleneck). Mas este argumento, em lugar de constituir uma solução para as contradições entre estimativas directas e indirectas, como sugerido nos exemplos anteriores, seria suficiente para rejeitar as estimativas indirectas de m que se deduzem dos valores de FST.
Os exemplos em Quercus (cf. secção seguinte), ao deixarem implícitas taxas de migração significativas entre populações separadas de centenas de quilómetros [Ducousso et al. 1993, Zanetto et al. 1994, Michaud et al. 1995, Elena-Rosselló et al. 1996, Toumi & Lumaret 1998, Jiménez et al. 1999], não são consistentes com o que actualmente se conhece da biologia da reprodução neste género (cf. parte II). Acrescente-se que, contrariamente ao que tem sido repetidamente recomendado, o número de loci é sempre bastante pequeno, geralmente faltando a referência ao número dos que não são polimórficos, dando assim a falsa impressão que a diversidade alélica apresentada é representativa do genoma [DeWoody & DeWoody 2005]. Também é sintomática a ausência de comentários sobre a dispersão de valores de locus para locus evidente em cada estudo, quando sob o pressuposto de neutralidade selectiva deveriam constituir estimativas de um só valor para todo o genoma.
Voltando ao estudo em Euphydrias editha, a rejeição da hipótese “seleccionista” — que o FST baixo em vários loci resultaria de selecção estabilizante, convergente entre subpopulações — baseia-se no julgamento de que esses valores FST são demasiado homogéneos entre si para a diversidade de pressões selectivas e mutacionais que deveriam actuar nos diferentes loci [Slatkin 1987]. Mas a realidade é que os valores de FST nesse estudo não são homogéneos: em 2 dos 7 loci considerados neutrais em Euphydrias editha, os valores de Nem desviam-se da média mais do triplo do respectivo erro-padrão. O pressuposto de neutralidade selectiva implica que, como amostragem “aleatória” do genoma, deveriam ser representativos dum único valor (que a média entre eles aproximaria), o que não parece verificar-se para os sete. Alternativamente, tem-se a perspectiva “seleccionista” de selecção estabilizante, em que quanto mais baixo o FST num locus, maior é o presumível efeito desse modelo de selecção (não o fluxo genético representado por Nem) a contrariar a divergência entre subpopulações nesse locus; dos 8 loci polimórficos estudados em Euphydrias editha, apenas o de maior FST, apesar de minoritário, poderia estar mais próximo de ser neutral (Nem = 0,6, enquanto pelos métodos directos seria 0,1).
O problema, para demonstrar o efeito da selecção, é que têm de evidenciar-se e mesmo quantificar-se para cada locus as componentes do fitness subjacentes aos resultados. A análise das distribuições genotípicas em 4 loci de esterases de Hordeum vulgare L. (numa metapopulação derivada de cruzamentos entre variedades representativas de todas as proveniências da espécie), em sucessivos pontos do ciclo de vida (mais precisamente, plântula-adulto-plântula), revelou oscilações das frequências no espaço de 1 geração, nalguns casos de grande magnitude [Clegg et al. 1978]. Estas oscilações em geral não estavam associadas a variações evolutivas (consideradas pelas diferenças após um intervalo de 10 gerações), e em diversos casos eram de grandeza muito superior a estas. Da análise concluiu-se que havia diferenças importantes de viabilidade e de fertilidade entre genótipos, embora não forçosamente associadas a estes, nem a loci a eles ligados proximamente: citando os autores, «o que é medido através dos loci marcadores é o fluxo selectivo transmitido através do genoma pela estrutura interdependente de cada distribuição multi-locus», ou seja, os loci mais directamente afectados por interacções selectivas arrastam consigo outros loci que de alguma maneira interagem com eles, e esta rede de interacções é de tal modo difusa que os marcadores genéticos analisados (neste caso, os loci das esterases) apenas servem para evidenciar que existe selecção, sem que se possa dizer como ela é transmitida dos loci onde ela se exerce mais directamente para esses marcadores [Ziehe & Müller-Starck 1991].
Há outras demonstrações deste fenómeno em loci enzimáticos, por exemplo no caso da fosfoglucose isomerase, referenciado anteriormente (cf. secção 1) e no acompanhamento dos padrões adaptativos associados a caracteres biométricos por uma parte da variação isoenzimática (um locus para esterase e outro para fosfoglucomutase) [Volis et al. 2003, 2005]. Mas revisitando os dados de Bacilieri et al. [1996] (figura 2.7 e texto respectivo), nota-se que entre fases sucessivas dum único ciclo de vida as frequências dos genes se alteram marcadamente; é de supor que elas oscilam em função das vantagens relativas de diferentes alelos no locus em diferentes fases do ciclo, o que invalida os cálculos feitos quanto à taxa de hibridismo entre as espécies em questão, como já discutido anteriormente (parte II secção D2, “Interpretação dos marcadores nucleares de hibridismo”). Mais: colocam em causa o pressuposto de neutralidade dos marcadores isoenzimáticos.
Este padrão de oscilações, evidenciando complementaridade entre alelos na determinação do fitness máximo, explica não só a existência de polimorfismos, mas também a não-divergência entre (sub)populações de origem comum, mesmo que quase isoladas entre si, pois as frequências dos diferentes alelos, desde que não muito sujeitas aos efeitos da deriva, acabam por convergir para valores semelhantes dumas para as outras, independentemente do fluxo genético que possa haver entre elas.
Ducousso et al. [1993] reviram as publicações de parâmetros populacionais relativos ao género Quercus, tendo realçado dois aspectos, que são confirmados na generalidade das revisões e estudos originais realizados entretanto em populações adultas [Sork et al. 1993b, Kremer & Petit 1993, Elena-Rosselló et al. 1996, Kanazashi et al. 1997a, Streiff et al. 1998, Chung et al. 2002, Jensen et al. 2003]: os valores de FIS rondando 0,1 medidos em várias populações de carvalhos americanos que, considerando a ausência de autopolinização, foram interpretados como indicadores de estratificação na reprodução, ou seja, do efeito de Wahlund; e os valores de FST consistentemente inferiores a 0,1, indicando uma relativa homogeneidade entre populações que foi interpretada, como é usual, pela existência de fluxos genéticos entre populações suficientes para impedir uma divergência pronunciada. Segundo os autores, o primeiro aspecto poderia relacionar-se com a dispersão de sementes preferencialmente a curta distância, levando a que árvores relativamente próximas sejam geneticamente aparentadas e, se estiverem mais representadas na descendência umas das outras, formando grupos subpopulacionais cuja diversificação genética resulta no efeito de Wahlund e o valor positivo de FIS — note-se que esta situação é longe de ser generalizada, como se demonstrou pelos valores de FIS em vários loci enzimáticos de Q. ilex, cujo afastamento do valor 0 praticamente correspondia ao erro estatístico [Michaud et al. 1995]. O segundo aspecto parece ligar-se à distância de dispersão através do pólen, que teria de atingir mais de 100 Km, assim colocando em contacto entre si povoamentos que possam estar praticamente isolados entre si no que respeita à dispersão através da semente; note-se que esse valor não é confirmado pelas análises mais recentes, e provavelmente a homogeneidade entre populações é mantida por selecção estabilizante nos diversos loci polimórficos analisados.
– Em Q. suber o valor médio de Ae obtido de 14 loci variou entre 1,24 e 1,31 na Península Ibérica, enquanto para os 9 loci polimórficos se acharam valores de FST entre 1 e 6% [Jiménez et al. 1999]; porém, noutros estudos nesta espécie a diferenciação entre populações traduziu-se em valores bastante mais elevados, FST entre 2 e 36% (13 loci polimórficos, Península Ibérica [Elena-Rosselló et al. 1996]) ou GST entre 3 e 18% (7 loci polimórficos, bacia ocidental do Mediterrâneo [Toumi & Lumaret 1998]), sendo incerto se estas diferenças se deveram à discrepância entre loci amostrados, talvez decorrente da utilização de folhas. Quanto à opinião desta diferenciação mais elevada dever-se à detecção, nalgumas populações, de genes presumivelmente de Q. ilex [Toumi & Lumaret 1998], contrasta com a omissão da mesma para os valores de GST entre 6 e 17% num estudo análogo em Q. ilex [Michaud et al. 1995].
– Os estudos em Q. petræa e Q. robur, na Europa, mostram que as duas espécies são bastante semelhantes em termos de polimorfismo e de diferenciação populacional: os valores médios de Ae (ou ν) oscilam entre 1,25 e 1,49, enquanto os valores de FST ou GST são bastante baixos, na ordem de 2 a 3% (os valores de δ tendem a estar perto de 7 a 9%) [Herzog 1996]. Trata-se dum elevado polimorfismo para loci enzimáticos (maior do que a tendência das perenes arbóreas e arbustivas [Hattemer 1991, Hamrick et al. 1979]), cada população evidenciando o mesmo fundo genético que as restantes da mesma espécie. A distância genética d0, entre populações da mesma espécie, é apenas um pouco inferior à que separa estas duas espécies [Herzog 1996], sugerindo-se daí uma elevada proximidade filogenética de ambas. Nestas duas espécies, a investigação dos genótipos de 11 loci, em árvores adultas para as quais foi avaliado o respectivo grau de vitalidade (avaliado por caracteres morfológicos), demonstrou que, apesar de não haver aparente redução do polimorfismo genético (ν) em árvores menos saudáveis, estas tinham um maior coeficiente de fixação F, ou seja, menor heterozigose [Hertel & Zarpel 1996].
– Gram & Sork [2001], a partir de amostras de 9 locais onde predominam Q. alba, Carya tomentosa Nuttell (Juglandaceæ) e Sassafras albidum (Nuttell) Nees (Lauraceæ), distando entre si quando muito de duas dezenas de quilómetros, seguiram um procedimento de análise para o conjunto dos loci enzimáticos estudados, compatível com estatística multivariada, em que para cada indivíduo se construiu um vector genotípico (cada elemento descrevendo um gene diferente, com valores de 0 a 1, desde a ausência à homozigose). Nas 3 espécies foi identificada uma forte influência da estrutura florestal (densidade do povoamento, idade, etc.) nos genótipos observados, em contraste com a aparente independência de características abióticas de cada povoamento (tipo de solo, ponto cardeal de exposição ao Sol). No caso de Q. alba, 10 genes de 5 loci serviram para evidenciar correlações entre genótipos (especialmente no alelo 2 duma fosfoglucose isomerase) e densidade de árvores, principalmente das da classe de maior diâmetro (DAP maior ou igual a 37 cm). Nas outras espécies, foram diferentes as correlações detectadas entre genótipos e estrutura florestal. As variáveis relacionadas com a estrutura florestal são elas mesmas, na opinião dos autores, «bioensaios do ambiente», tendo de procurar-se a um nível de refinamento muito maior as causas primárias da variação a esse nível, às quais então se poderia atribuir um papel selectivo.
– O estudo de 18 populações de Q. petræa abrangendo várias regiões da distribuição desta espécie [Zanetto et al. 1993] revelou diferenças interessantes entre loci e entre regiões geográficas, embora difíceis de interpretar. Para 6 loci enzimáticos polimórficos e segundo as populações, foi obtido um valor He médio entre 0,29 e 0,40 (Ae entre 1,41 e 1,67), mas quando se refizeram os cálculos separadamente para 3 loci de enzimas considerados do metabolismo primário — glutamato-oxaloacetato transferase (GOT), fosfoglucose isomerase (PGI) e fosfoglucomutase (PGM) — os valores médios de He eram mais baixos, enquanto para os restantes, considerados do metabolismo secundário — fosfatases ácidas (ACP), diaforase (DIA), menadiona reductase (MR) — eram mais elevados. Os valores de GST variaram entre 2 e 4%, sem aparentemente distinguirem as duas categorias de loci, mas para os loci de metabolismo secundário na parte central da distribuição (entre França, Bélgica e Alemanha) eram substancialmente mais baixos (perto de 1%). Embora a interpretação tenha de ficar em aberto, sugeriu-se que se encontraria uma correspondência mais directa entre diferenciação genética (GST) e geográfica nas actividades enzimáticas codificadas por este grupo de loci, já de si com maior riqueza alélica, mas sem se poder definir se seria adaptativa ou meramente estocástica (deriva genética).
– A comparação entre dois povoamentos na bacia do Reno de Q. robur contíguos entre si, um deles sofrendo alagamento todos os anos [Herzog & Krabel 1999], com base em 17 loci (monomórficos e polimórficos), revelou uma distância genética d0 de 7,4%. Esta diferenciação entre os dois povoamentos — se bem que pequena — devia-se sobretudo a 3 loci (PGD-A, EST-A e SKDH-A, respectivamente para 6-fosfogluconato desidrogenase, esterase e chiquimato desidrogenase), sendo notável um alelo de EST-A, que costuma ser raro noutras regiões, aqui ter frequências bastante elevadas: 4,5% no povoamento que não era alagado e de 20% no que era. O facto das frequências deste alelo nas descendências obtidas a partir de sementes destes povoamentos baixarem para 3% e subirem para 29%, respectivamente, era sugestivo duma pressão selectiva associada à ocorrência de alagamentos. Este seria, além disso, mais um exemplo de substanciais flutuações de frequências durante o ciclo de vida, que por analogia com casos estudados mais detalhadamente (analisado na secção anterior) podem estar associados a pressões selectivas. Aliás, variações noutros loci entre adultos e descendências do mesmo povoamento contribuíram para uma distância d0 entre gerações do mesmo povoamento quase tão grande como entre povoamentos.
– A disparidade de características genéticas entre adultos e descendências da mesma população também foi observada noutro estudo em Q. robur na bacia do Reno, mas desta vez abrangendo 15 regiões bastante afastadas entre si, e referenciando-se apenas a 6 loci polimórficos [Gehle 1999]. Concluiu-se dessa disparidade que não haveria equilíbrio genético, fosse ele neutral ou selectivo, mas de maior importância foi a consequência prática de revelar-se impossível correlacionar materiais de propagação com a sua origem, mesmo utilizando a informação de vários loci.
– A amostragem de 56 populações de Q. ilex abrangendo grande parte da distribuição actual da espécie [Michaud et al. 1995] mostrou que apenas 7 loci eram polimórficos (ao contrário de quase todos os outros estudos europeus com Quercus, neste usaram-se folhas das árvores adultas). O valor médio de He era de 0,22, mas era apenas de 0,13 no que se consideraram populações “marginais” (biótopos extremos para a espécie, em termos de demasiada geada ou excessiva humidade; a única de Portugal, na Ria Formosa, pertencia a este grupo). O valor de GST, cerca de 10%, encontra paralelo noutros estudos de loci enzimáticos a esta escala geográfica [Sork et al. 1993b]. Usando análise de componentes principais (PCA) para os resultados em 5 desses loci de 55 das populações, obtiveram-se 3 agrupamentos estatísticos de 42 populações, cujo significado não é fácil de discernir, pois não era aparente qualquer relação com a diferenciação taxonómica (que também é ecológica) entre as subespécies ilex e rotundifolia, nem com o isolamento geográfico definido no próprio artigo. Para além do número limitado de loci polimórficos disponíveis é de notar a ênfase em métodos estatísticos valorizando alelos raros, o que é impróprio tendo em conta a reduzida amostragem por população (rondando 30 indivíduos).
Face ao conhecimento presente, onde os marcadores de DNA trouxeram uma nova luz sobre os mais diversos aspectos da biologia das populações de Quercus, a análise isoenzimática fica como uma técnica do passado, que trouxe pouca informação utilizável. Como ilustrado nos estudos aqui resumidos, isso deve-se em parte ao desenho da amostragem, em parte ao tratamento dos dados, e no geral à insistente presunção de neutralidade selectiva destes marcadores.
1) Teoria para os padrões isoenzimáticos
Cada amostra submetida a electroforese é uma mistura de enzimas, e a revelação de cada
actividade enzimática permite, com base na sua especificidade catalítica, evidenciá-la em relação à cor
do gel. Cada enzima migra dentro do gel paralelamente ao eixo formado entre os pólos eléctricos da
electroforese e, ao fazer-se a respectiva revelação, para cada amostra pode surgir uma ou mais áreas
coradas (“bandas”
![]() ) que evidenciam enzimas de mobilidades diferentes mas com a mesma actividade
enzimática (isoenzimas).
) que evidenciam enzimas de mobilidades diferentes mas com a mesma actividade
enzimática (isoenzimas).
Partindo do princípio que cada gene codifica um polipéptido, o número de diferentes bandas duma actividade enzimática por indivíduo depende:
i) do número de loci que a codificam;
ii) do número de alelos resolúveis após electroforese em cada locus;
iii) do padrão de associação entre polipéptidos para formação de cada enzima activo, seja entre os codificados no mesmo locus ou entre os de loci diferentes.
O caso mais simples é o dos enzimas monoméricos (constituídos apenas por um polipéptido) codificados por apenas um locus; assim, cada indivíduo diplóide apresenta apenas 1 ou 2 bandas, consoante seja homozigótico ou heterozigótico, respectivamente; a complexidade aumenta de diversas maneiras, seja por haver loci duplicados para a mesma actividade que se expressam separadamente no mesmo material em análise (por exemplo, uma actividade no citosol e outra nos cloroplastos), seja pela associação entre polipéptidos de loci diferentes no mesmo compartimento celular. Quando se trata de actividades enzimáticas com um espectro de actividade mais amplo, como é o caso de certas hidrolases, peroxidases, etc. [Johnson 1979], o número de loci implicados pode ser elevado e consequentemente encontrarem-se muitas bandas.
Outro factor a levar-se em conta em certos casos é a modificação dos polipéptidos com grupos funcionais que lhes alteram as propriedades de migração no gel: fosfatos, lípidos, etc. [Poly 1997]. As bandas adicionais que resultam não representam genes adicionais, mas aumentam a complexidade da interpretação genética.
Ilustração das interpretações genéticas
Formalmente, a interpretação dos zimogramas obtidos após electroforese faz-se a partir de cruzamentos controlados, com recurso às leis de Mendel, ao conhecimento da ploidia das células donde se extraíram os enzimas e da estrutura quaternária destes. Não existindo esse conhecimento para cada sistema enzimático na espécie em estudo, um ponto de partida defensável é supor-se que espécies congenéricas têm a mesma estrutura quaternária.
Em regra, para cada alelo ao qual se atribui uma banda resolvida define-se uma distância de migração no gel, tipificada pelos respectivos homozigóticos nesse locus. Nos heterozigóticos, aparecem as posições para os dois alelos, às quais pode ou não juntar-se uma ou mais posições intermédias, dependendo de poderem formar-se estruturas quaternárias mistas. Os exemplos na figura 2.10 ilustram os padrões básicos de variação com segregação de apenas 2 alelos (a/a'), usando como exemplos 4 enzimas com diversas estruturas quaternárias.
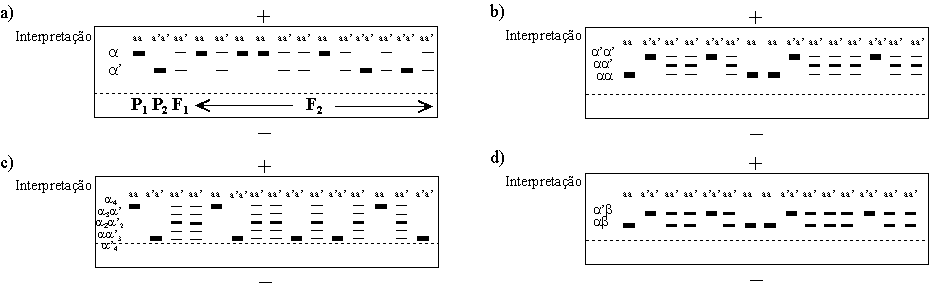 Figura 2.10 — Exemplo fictício dos resultados da análise por electroforese de 4 actividades enzimáticas dum
mesmo conjunto de amostras. Como se indica na primeira imagem, em cada “zimograma” estão ordenadas as
seguintes amostras, da esquerda para a direita: progenitores P1 e P2 (homozigóticos), a respectiva F1 e 12
indivíduos da F2 resultantes de selfing na F1. Em todos os casos há segregação num só locus (alelos a/a', péptidos
α/α' respectivamente), sendo dada a interpretação genotípica para cada amostra no alto do respectivo zimograma,
mostrando-se à esquerda, alinhadas com as respectivas bandas, as fórmulas peptídicas correspondentes. Os sinais
+ e – dão a orientação do gel durante a electroforese em relação ao ânodo e ao cátodo, respectivamente. A linha
horizontal a tracejado assinala a origem de migração dos enzimas. a) Enzima monomérico; b) enzima
homodimérico (constituído por dois polipéptidos codificados no mesmo locus); c) enzima homotetramérico (idem,
4 polipéptidos); d) enzima heterodimérico (dois polipéptidos α e β, codificados por dois loci, estando o locus
segregante a/a' ligado ao locus da alínea b (recombinação nula dentro deste conjunto de amostras).
Figura 2.10 — Exemplo fictício dos resultados da análise por electroforese de 4 actividades enzimáticas dum
mesmo conjunto de amostras. Como se indica na primeira imagem, em cada “zimograma” estão ordenadas as
seguintes amostras, da esquerda para a direita: progenitores P1 e P2 (homozigóticos), a respectiva F1 e 12
indivíduos da F2 resultantes de selfing na F1. Em todos os casos há segregação num só locus (alelos a/a', péptidos
α/α' respectivamente), sendo dada a interpretação genotípica para cada amostra no alto do respectivo zimograma,
mostrando-se à esquerda, alinhadas com as respectivas bandas, as fórmulas peptídicas correspondentes. Os sinais
+ e – dão a orientação do gel durante a electroforese em relação ao ânodo e ao cátodo, respectivamente. A linha
horizontal a tracejado assinala a origem de migração dos enzimas. a) Enzima monomérico; b) enzima
homodimérico (constituído por dois polipéptidos codificados no mesmo locus); c) enzima homotetramérico (idem,
4 polipéptidos); d) enzima heterodimérico (dois polipéptidos α e β, codificados por dois loci, estando o locus
segregante a/a' ligado ao locus da alínea b (recombinação nula dentro deste conjunto de amostras).Na figura 2.11, ilustram-se mais alguns exemplos com séries alélicas ou interacções mais complexas.
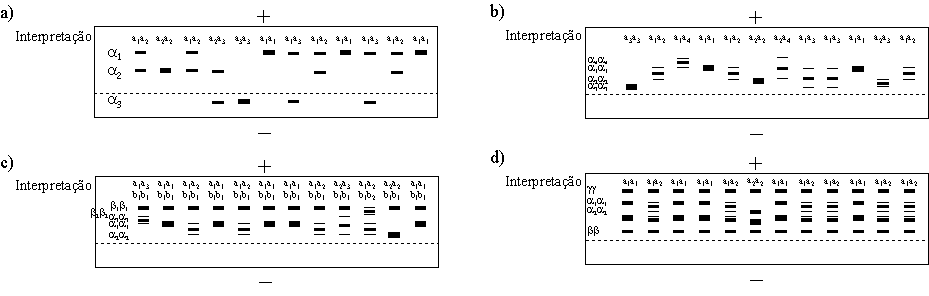 Figura 2.11 — Exemplo de 4 “zimogramas” fictícios respeitantes a 12 amostras de indivíduos sem parentesco entre
si. Convenções como explicado na legenda da figura anterior, embora só se ilustrem algumas fórmulas peptídicas
(à esquerda em cada figura). a) Enzima monomérico, alelos a1/a2/a3 (note-se que o aloenzima codificado por a3
migra para o cátodo); b) enzima homodimérico, alelos a1/a2/a3/a4 (convenciona-se aqui designar por a1 o alelo mais
frequente, apesar de não ser o que migra mais ou menos dentro do gel); c) dois isoenzimas homodiméricos
expressos em diferentes compartimentos celulares (polipéptidos α e β), alelos a1/a2/a3 e b1/b2; d) sistema de
isoenzimas diméricos codificados em 3 loci, dos quais apenas o a1/a2 é polimórfico — os polipéptidos α e β
localizam-se no mesmo compartimento celular de modo que se podem formar heterodímeros αβ (o polipéptido γ
localiza-se noutro compartimento).
Figura 2.11 — Exemplo de 4 “zimogramas” fictícios respeitantes a 12 amostras de indivíduos sem parentesco entre
si. Convenções como explicado na legenda da figura anterior, embora só se ilustrem algumas fórmulas peptídicas
(à esquerda em cada figura). a) Enzima monomérico, alelos a1/a2/a3 (note-se que o aloenzima codificado por a3
migra para o cátodo); b) enzima homodimérico, alelos a1/a2/a3/a4 (convenciona-se aqui designar por a1 o alelo mais
frequente, apesar de não ser o que migra mais ou menos dentro do gel); c) dois isoenzimas homodiméricos
expressos em diferentes compartimentos celulares (polipéptidos α e β), alelos a1/a2/a3 e b1/b2; d) sistema de
isoenzimas diméricos codificados em 3 loci, dos quais apenas o a1/a2 é polimórfico — os polipéptidos α e β
localizam-se no mesmo compartimento celular de modo que se podem formar heterodímeros αβ (o polipéptido γ
localiza-se noutro compartimento).A análise isoenzimática em plantas tem como obstáculo frequente a perda de actividade dos enzimas dos tecidos logo que se faz a extracção. O problema reside principalmente na presença de taninos e outros compostos fenólicos, mas também há referências à adsorção das proteínas a estruturas macromoleculares e supramoleculares ionizadas (facilitada pela queda da força iónica no momento da rotura dos vacúolos), como sejam ácidos nucleicos, fibras das paredes celulares (pectinas), ou membranas celulares [Loomis & Bataille 1966, Loomis 1974, Skopes 1987]. Outro factor a ter em conta é a abundância de terpenos e outros metabolitos secundários (ácidos resiníferos, carbonilos, isoflavonas, comarinas, carotenóides, etc.) inibidores das actividades enzimáticas [Loomis 1974].
Os taninos e outros fenóis localizam-se nos vacúolos das células fotossintéticas e só entram em contacto com os restantes constituintes celulares por ruptura do tonoplasto, como por exemplo em tecidos danificados; uma vez libertados, formam complexos insolúveis com as proteínas [Loomis & Bataille 1966, Loomis 1974] e, quando em contacto com o dioxigénio e a pH neutro ou alcalino, ou ainda pela acção das fenoloxidases (tirosinases) presentes no citosol [Anderson 1968], ionizam-se em quinonas que formam espontaneamente ligações covalentes com as proteínas e as inactivam [Loomis & Bataille 1966, Anderson 1968, Loomis 1974]. A maior ou menor quantidade de taninos e outros fenóis, e a actividade das fenoloxidases, têm grande relevância fisiológica nomeadamente na defesa das plantas terrestres contra patogénios, de certo modo análoga à formação do trombo na circulação sanguínea, pois resulta num ambiente desfavorável ao crescimento de patogénios nos tecidos danificados ou senescentes, isolando-os dos tecidos sãos [Anderson 1968, Okey et al. 1997].
Por isso, a análise isoenzimática em plantas tem de realizar-se ao abrigo destes factores de inactivação. Durante um lapso de tempo prolongado só se conseguiam resultados onde o teor em taninos e outros fenóis fosse baixo, seja nas folhas de certas espécies ou cultivares, ou noutros órgãos com essa característica, como por exemplo gemas ou botões foliares [May1991], ou ainda estudando actividades enzimáticas que não eram afectadas pelos taninos [Mitton et al. 1977]. Embora se conhecesse a acção protectora das actividades enzimáticas de diversos reagentes, a maneira de combiná-los eficientemente parece só ter começado com o estudo de Kelley & Adams [1977] para a extracção a partir das folhas maduras duma conífera. Envolvia o emprego duma solução tamponada a pH 7, contendo uma alta concentração de antioxidante, um inibidor das fenoloxidases, e outros compostos complexantes de taninos e de fenóis simples, além do uso de azoto líquido (que protege pela baixa temperatura, facilita a maceração das folhas e cria uma atmosfera não-oxidante), e da inclusão dum conservante e um passo de extracção com álcoois. Esse estudo demonstrava de maneira convincente que só a utilização simultânea de todos esses ingredientes permitia obter zimogramas de qualidade para três actividades enzimáticas que serviam de teste [Kelley & Adams 1977].
Nesse trabalho de Kelley e Adams desembocava pouco mais duma década de sugestões sobre o modo de ultrapassar as contingências da extracção dos enzimas em plantas antes da electroforese. Na tabela 2.6 resume-se uma parte substancial deste conhecimento, coligida para o presente trabalho.
A aplicação destes princípios, com algumas modificações para tornar o procedimento mais expedito e mesmo assim ser adequado para várias espécies de coníferas, foi apresentada por Mitton et al. [1979]. Mas nem sempre se considera compensador investir tempo para encontrar condições de extracção que mantenham o máximo número de actividades enzimáticas em solução para cada espécie ou órgão a analisar, além de muitos considerarem criticável a complexidade das soluções usadas na extracção, ela própria um potencial factor de inactivação dalguns enzimas [Soltis et al. 1983, Kephart 1990, May 1991, Bult & Kiang 1993]. Talvez por isso se encontre nos anos mais recentes, por exemplo nos Quercus, alguma predominância de meios relativamente simples e a preferência por órgãos com menos taninos, que são conservados a temperaturas muito baixas até à extracção, a qual se realiza imediatamente antes da electroforese [Yacine & Lumaret 1989, von Wühlisch & Muhs 1995, Nóbrega 1997, Jiménez 2001].
Tabela 2.6 — Lista das principais opções relativas aos diversos componentes da solução utilizada para extracção de isoenzimas.
Função |
Opções |
Comentários |
Referênciasa |
Substâncias tampão |
tris, fosfato |
pH neutro a alcalino |
1, 2, 3, 4 |
tris-maleato |
pH neutro |
4, 5 |
|
maleato, histidina |
pH ácido a neutro; histidina complexa catiões divalentes |
6, 7 |
|
Antioxidantes tiol (neutralizam radicais livres nos grupos tiol reduzidos das proteínas, e podem ter outras funções) |
glutationa, cisteína |
não servem para conservação (máximo 24 horas), também são quelantes do Cobre (cf. DIECA) e complexantes de quinonas |
4, 7, 8 |
2-mercaptoetanol |
muito utilizado apesar do mau cheiro e de não servir para conservação (máximo 24 horas), também quelante do Cobre (cf. DIECA) |
3, 4, 6, 7, 9 |
|
ditiotreitol (DTT) |
reagente “desenhado” para substituir os três anteriores na função antioxidante, permite conservação prolongada |
4, 7 |
|
tioglicolato, mercaptobenzotiazol |
revertem a oxidação dos fenóis em quinonas |
8 |
|
Antioxidante não-tiol |
ascorbato |
neutraliza radicais livres e regenera grupos tiol reduzidos |
6 |
Inibidores de tirosinases |
dietilditiocarbamato (DIECA) |
quelante do Cobre (cofactor das tirosinases), complexa quinonas |
8 |
azida, cianeto |
competidores do dioxigénio para o centro activo; muito venenosos |
|
|
germanato, tetraborato |
inibidores competitivos, também complexam alguns fenóis e glúcidos |
2, 11 |
|
metabissulfito |
inactivador da tirosinase, reage com polifenóis e quinonas, mas pode ser contraindicado, pois reage com muitos grupos químicos, entre os quais pontes dissulfureto; protege da oxidação |
8, 9 |
|
Adsorventes de fenóis complexos (taninos) e simples |
polivinilpirrolidona (PVP) |
macromolécula que mimetiza a ligação peptídica, que é onde se ligam os taninos |
1, 6 , 8, 9 |
polivinilpolipir-rolidona (PVPP, Polyclar-AT) |
forma insolúvel do anterior |
6, 9 |
|
acrilamidas |
complementam PVP para derivados do catecol, tirosina |
6, 11 |
|
sulfato de protamina, Tween 80 |
dissociam complexos não covalentes entre proteínas e fenóis |
9, 10 |
|
fenoxietanol |
complexa fenóis simples |
2 |
|
Outros adsorventes |
EDTA |
quelante de catiões divalentes (cf. Mg2+), também inibidor de proteases |
4, 7, 12 |
Albumina do soro de bovino (BSA) |
adsorvente de polifenóis, polianiões, de ácidos gordos e outros lípidos, etc.; também contribui para uma concentração de proteína adequada para a conservação |
4, 6, 7, 9 |
|
dextrano, Ficoll |
adsorventes de catiões |
4 |
|
resinas de polistireno poroso (Amberlite XAD-2 e XAD-4) |
removem detergentes e terpenos, mas o seu uso em coluna não é prático para processar amostras em grande número |
9 |
|
Estabilizadores e solventes |
Mg2+ |
estabilizador de membranas celulares; a sua remoção pelo EDTA facilita a permeabilização das membranas (como fazem os detergentes) |
4 |
álcoois |
separação de lípidos inibidores |
6, 11, 12 |
|
etileno glicol, glicerol |
redução da actividade da água, anticongelantes |
7, 13 |
|
Detergentes |
Triton X-100, CHAPS, Tween 80, sais da bílis |
não-iónicos excepto CHAPS (aniónico e catiónico); pelo menos o Tween 80 reverte a interacção dos taninos com as proteínas |
9, 12, 14 |
Osmolaridade e força iónica |
sacarose, manitol, sorbitol, KCl |
regulação da osmolaridade |
4, 7 |
KCl |
regulação da força iónica |
7 |
|
Inibidores |
NaF |
inibe glicólise, evitando os efeitos da fermentação láctica no pH |
7 |
de proteases (diversas classes) |
protecção a prazo |
7 |
|
Conservante |
DMSO |
protecção a prazo |
11 |
a 1 [Kephart 1990] 2 [Mitton et al. 1979] 3 [Soltis et al. 1983] 4 [Theimer 1983] 5 [Bult & Kiang 1993] 6 [Loomis & Bataille 1966] 7 [Skopes 1987] 8 [Anderson 1968] 9 [Loomis 1974] 10 [Goldstein & Swain 1965] 11 [Kelley & Adams 1977] 12 [Penefsky & Tzagoloff 1971] 13 [Gekko & Timasheff 1981] 14 [Hjelmeland 1980]
3) Princípios gerais da histoquímica
A histoquímica baseia-se nas propriedades químicas dos diversos grupos de moléculas biológicas para as diferenciar entre si. O resultado que se espera duma reacção histoquímica é a acumulação dum composto corado que permanece, geralmente pela sua insolubilidade, suficientemente próximo das moléculas a detectar para assinalar a sua localização com exactidão, e precisão, adequadas à escala de resolução a que se observa.
No caso dos enzimas, a propriedade química específica que se explora é o tipo de actividade catalítica, definida no respectivo código preconizado pela Comissão de Nomenclatura da International Union of Biochemistry and Molecular Biology (Enzyme Commission, EC) [Lehninger 1975, BRENDA]. A visualização de cada actividade enzimática faz-se onde, a partir dum substrato apropriado que é fornecido, se forma o produto resultante da sua catálise — seja pela deposição in situ desse produto, dum seu derivado, ou de qualquer subproduto dele dependente, e cuja cor (ou fluorescência) contraste com localizações onde esse produto não se forma.
Particularidades da revelação histoquímica após electroforese
Normalmente, a revelação de actividades enzimáticas após uma electroforese é feita pela catálise específica no próprio gel, de modo a que o local onde existir uma molécula com a actividade a detectar seja assinalado pelo depósito visualizável formado em resultado dessa catálise; na maior parte dos casos a revelação é por contraste positivo, isto é, visualizam-se as actividades enzimáticas pela formação de produtos corados sobre um fundo mais claro (em regra branco ou transparente, segundo se trata de amido ou poliacrilamida), porém há exemplos de contraste negativo (fundo corado, produto mais claro).
Algumas revelações realizam-se, não por imersão do gel na solução de revelação, mas numa camada contendo os ingredientes da reacção que se forma sobre a superfície do gel. Essa camada pode ser de papel de filtro ou de agarose, permitindo concentrar os mesmos ingredientes num volume mais reduzido, aumentando a eficiência das reacções para um igual consumo de reagentes [Soltis et al. 1983, Allen et al. 1984, May 1991, Acquaah 1992].
A visualização é macroscópica, e por isso exige uma concentração relativamente elevada de enzima para que seja perceptível. Este aspecto limita bastante o número de enzimas que podem ser demonstrados nas amostras — por exemplo, é comum actividades abundantes num órgão serem indetectáveis noutros órgãos. Além disso, a distinção entre isoenzimas reside na nitidez das diferenças de migração a partir da origem durante a electroforese, o que só é possível se cada isoenzima tiver uma taxa de migração muito homogénea, traduzindo-se numa zona de coloração estreita (a “banda”), que favorece a sua resolução e, também, a intensidade da coloração.
A matriz dos géis de amido é tão permeável às macromoléculas que as diferenças de migração residem essencialmente nas diferenças de ponto isoeléctrico e do pH escolhido [Acquaah 1992]. Verifica-se empiricamente que o sistema de soluções-tampão usado durante a electroforese pode interferir com a visualização de vários enzimas: em primeiro lugar, o pH do gel tem de ser adequado para optimizar a separação entre diferentes isoenzimas; em segundo lugar, a química do sistema-tampão pode inviabilizar a nitidez das bandas (presumivelmente por desnaturação parcial durante a electroforese) e até inibir uma actividade enzimática [Acquaah 1992]. Embora existam listas de sistemas de separação para cada actividade enzimática a ensaiar, sugeridas para os diferentes grupos de organismos [May 1991], não é incomum verificar-se que a mesma actividade enzimática é analisada na mesma espécie com sistemas diferentes consoante o laboratório [Micales et al. 1986, Selander et al. 1986, Kephart 1990, May 1991, Bult & Kiang 1993]. A experimentação com diferentes alternativas é um passo preliminar geralmente incontornável.
O mesmo trabalho de experimentação de alternativas pode ser necessário, ocasionalmente, também para a revelação de algumas actividades enzimáticas, seja no pH do tampão ou até na escolha do substrato mais adequado.
Finalmente, ao contrário do que é norma na histoquímica para microscopia, as actividades enzimáticas não são fixadas in situ após a electroforese, difundindo e decaindo rapidamente. Por isso é indispensável proceder à revelação imediatamente.
4) Tipos de reacções de revelação
Entre a catálise realizada in situ e a correspondente visualização podem ou não intercalar-se processos mais ou menos indirectos, e em função disso (não obstante poderem adoptar-se outros critérios, cf. Gabriel [1971], Heeb & Gabriel [1984], Gabriel & Gersten [1992]) definem-se aqui 3 classes de sistemas de revelação após a electroforese: a visualização directa do produto de catálise, o uso dum reagente de captura para precipitar o produto da reacção, e o encadeamento de reacções que conduzem à formação dum precipitado corado.
Visualização directa do produto da catálise

(S: substrato da catálise; E: actividade a detectar; P: produto da
catálise; V: composto visualizado).
O produto da reacção é a entidade visualizada. Globalmente designados como métodos autocrómicos [Gabriel 1971, Heeb & Gabriel 1984, Gabriel & Gersten 1992], segundo o tipo de visualização podem dividir-se em 2 grupos:
Produto visível por contraste negativo em luz normal, exemplos: para a CAT (catalase, EC 1.11.1.6), a reacção do tiossulfato de sódio com KI produz I2, que cora o amido de negro-azulado, a servir de fundo excepto onde a produção de dioxigénio (O2) pela catálise inibe a formação desse I2 [Gabriel 1971, May 1991, Acquaah 1992]; para a SOD/TO (superóxido dismutase/tetrazólio oxidase, EC 1.15.1.1), a foto-redução espontânea de NAD/PMS traduz-se na redução do tetrazólio NBT e consequente escurecimento do fundo (cf. encadeamento de reacções), excepto onde se forma O2 pela catálise, que inibe essa foto-redução [Micales et al. 1986, May 1991, Gabriel & Gersten 1992, Acquaah 1992].
Produto autofluorescente sob luz UV (NADH, derivados de naftol ou de 4-metilumbeliferil, MeU), exemplo: EST-F (esterases, EC 3.1.1.2), o substrato é MeU-acetato ou MeU-butirato, e a hidrólise liberta o MeU fluorescente [Weder & Kaiser 1995, May 1991].
Combinação do produto da catálise com um reagente de captura
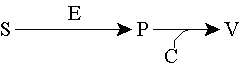 (C: reagente de captura)
(C: reagente de captura)
O produto da reacção forma com um reagente de captura um composto insolúvel que é visualizado. Grande parte destas reacções envolvem moléculas da família dos diazónios, exemplos: para a AAT/GOT (aspartato aminotransferase/ glutamato-oxaloacetato transaminase, EC 2.6.1.1) o substrato é natural (aspartato + α-cetoglutarato), formando-se oxaloacetato que é “capturado” por um diazónio, geralmente o Fast Blue BB [Gabriel 1971, Soltis et al. 1983, May 1991, Acquaah 1992, Bult & Kiang 1993]; para a ACP (fosfatase ácida, EC 3.1.3.2), o substrato é não-natural (α-naftil fosfato), e a captura faz-se entre o naftilo, formado após a hidrólise do éster de fosfato, e um diazónio como Fast Garnet ou Fast Black K [Heeb & Gabriel 1984, Micales et al. 1986, May 1991, Acquaah 1992, Bult & Kiang 1993]. Derivados de naftilo são usados para diversas hidrolases.
A formação de precipitados incolores, mas visíveis em géis transparentes (de poliacrilamida, nomeadamente), faz-se por acoplamento dum produto da catálise (fosfato, pirofosfato, ou CO2) com Cálcio na solução, exemplos: AKP (fosfatase alcalina, EC 3.1.3.1) usando p-nitrofenil fosfato e 10 mM Ca2+ a pH 10, PyrDC (piruvato descarboxilase, EC 4.1.1.1) usando 2 mM Ca2+ [Nimmo & Nimmo 1982, Heeb & Gabriel 1984, Gabriel & Gersten 1992].
A detecção das PER (peroxidases, EC 1.11.1.7), que produzem O2, faz-se pela oxidação de 3-amino-9-carbazol, precipitando-o [May 1991, Acquaah 1992].
Encadeamento de reacções a partir de cofactores da catálise
Em termos gerais, um dos produtos da reacção é o substrato que serve de ponto de partida para outras reacções, enzimáticas ou não, que resultam mais ou menos indirectamente na formação do composto de visualização.
Quando um dos produtos da reacção é NADH ou NADPH, transfere os seus equivalentes redutores para um tetrazólio, geralmente o MTT — abreviatura para brometo de 3-(4,5-dimetil-2-tiazolil)-2,5-difenil-2H-tetrazólio — que na forma reduzida (formazano) é de cor intensa e insolúvel. O encadeamento de reacções é não-enzimático e envolve dois ciclos redox, um repondo a forma oxidada do coenzima, outro envolvendo metossulfato de fenazina (PMS), que transfere os equivalentes redutores do produto da catálise enzimática para o tetrazólio:
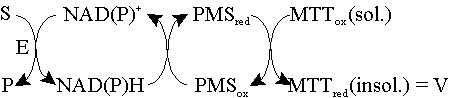
Outro tetrazólio utilizado, embora muito menos que o MTT, é o NBT (Nitro Blue Tetrazolium). São exemplos deste tipo de revelação dependentes do NAD as actividades ADH (álcool desidrogenase, EC 1.1.1.1), GAPD (gliceraldeído 3-fosfato desidrogenase, EC 1.2.1.12), LDH (lactato desidrogenase, EC 1.1.1.27), MDH (malato desidrogenase, EC 1.1.1.37), etc.; entre as dependentes do NADP, a G6PD (glucose 6-fosfato desidrogenase, EC 1.1.1.49), IDH (isocitrato desidrogenase, EC 1.1.1.42), ME (enzima málico, EC 1.1.1.40), PGD (6-fosfogluconato desidrogenase, EC 1.1.1.44), etc..
Uma modificação do encadeamento acima emprega-se nas actividades que utilizam NAD(P)H (reacção inversa), transferindo os equivalentes redutores para um substituto da forma oxidada de PMS, 2,6-diclorofenol indolfenol (DCPIP) ou alternativamente menadiona, daí passando-os para o MTT. Exemplo: DIA/MenR (diaforase/menadiona reductase, EC 1.6.4.3 ou 1.6.99.–), que utiliza NADH [May 1991, Acquaah 1992, Bult & Kiang 1993].
Um outro tipo de encadeamento redox foi proposto para a revelação de fosfo-hidrolases, em que o produto (ortofosfato) reduz o ácido molíbdico, transferindo-se o equivalente redutor através duma tetrabase (em alternativa à benzidina) para amónia e permanganato, formando um precipitado azul [Lomholt 1975].
Nesta classe de sistemas de revelação incluem-se os encadeamentos que envolvem reacções enzimáticas (catalizadas por enzimas incluídos na solução de reacção), no que se designa por acoplamento enzimático. A visualização faz-se a partir da actividade dum enzima indicador, o qual depende mais ou menos indirectamente da actividade inicial que se pretende detectar, e cuja posição fica marcada no gel. Aqui pode haver encadeamentos mais ou menos complexos, a maior parte conduzindo a um enzima indicador donde resulte a produção de NADH ou NADPH e consequente depósito de formazano — por exemplo, as actividades que produzem glucose 6-fosfato (designadamente a PGI-fosfoglucose isomerase, EC 5.3.1.9, a PGM-fosfoglucomutase, EC 2.7.5.1, e a HXK-hexoquinase, EC 2.7.1.1) são reveladas por encadeamento com a G6PD, a ACO (aconitase, EC 4.2.1.3) com a IDH, a FUM (fumarase, EC 4.2.1.2) com a MDH, a PK (piruvato quinase, EC 2.7.1.40) com a LDH, a TPI (triose fosfato isomerase, EC 5.3.1.1) com GAPD, etc. [Micales 1986, May 1991, Acquaah 1992].
Há actividades enzimáticas cujos produtos são detectados através dum múltiplo encadeamento enzimático. É o caso das PEP (peptidases, EC 3.4.11.– ou 3.4.13.–), onde se usa PER como indicador mas cujo substrato (H2O2) é produzido por uma L-aminoácido oxidase (AAO) [May 1991]:
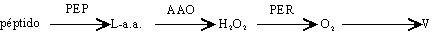
Outro exemplo é o da MPI (manose 6-fosfato isomerase, EC 5.3.1.8), cujo produto imediato é a fructose 6-fosfato, a partir da qual, através da catálise pela PGI e pela G6PD, produz NADPH que pode visualizar-se pela oxidação dum tetrazólio [May 1991].
D — Comparação genérica com outros métodos de análise molecular
Ressalvando a possibilidade teórica de sequenciar todo o genoma de cada indivíduo, os métodos disponíveis produzem genótipos que constituem uma amostragem da informação presente no seu genoma, e a representação que se consegue desse potencial informativo é diferente consoante as abordagens metodológicas que são elegidas. É assim que os descritores genotípicos dum indivíduo constituem “marcadores” do seu genoma que, na medida do possível, não se devem afastar de critérios ideais, como sejam: distribuição homogénea por todos os grupos de ligação, representação equilibrada das funções metabólicas existentes, perda de informação se possível nula em relação à variação ao nível da sequência nucleotídica, e, dentro dum certo contexto, neutralidade evolutiva. Outra ordem de critérios tem a ver com a sua aplicação prática, a analisar mais adiante para o hibridismo entre sobreiro e azinheira: facilidade de execução, reprodutibilidade, e economia de meios (nomeadamente, o número de indivíduos que podem ser analisados com os recursos e tempo disponíveis).
1) Fenótipos moleculares não-enzimáticos
Na análise do DNA, dois métodos permitem fazer uma amostragem da informação genómica teoricamente próxima do ideal, já que por princípio os marcadores obtidos são em grande número e distribuem-se aleatoriamente por todos os grupos de ligação, e além disso não estão, pelo menos a priori, associados a regiões com uma determinada função genética: os marcadores de RAPD (random amplified polymorphic DNA) e dos AFLP (amplified fragment length polymorphisms). Ambos se baseiam na utilização de primers de PCR arbitrários e produzem, para cada indivíduo, um grande número de fragmentos de DNA amplificado, de que uma proporção relevante é polimórfica. Contudo, trata-se maioritariamente de variações de presença/ausência de cada marcador, de tal modo que em cada locus os heterozigóticos não se distinguem dos homozigóticos, e é em geral bastante problemática a possibilidade de haver mais do que um alelo de presença (mas com diferentes tamanhos de fragmento) sejam tratados como não-alélicos. É necessário ter em conta a possibilidade dum ou mais marcadores ser citoplásmico em vez de nuclear [Mariette et al. 2002]. Além disso, o RAPD é na prática difícil de reproduzir entre laboratórios [Jones et al. 1997], e os AFLP, se bem que altamente reprodutíveis, requerem maior perícia e são especialmente sensíveis, em termos de reprodutibilidade, à maneira como é extraído o DNA [Jones et al. 1997, Chavarriaga-Aguirre et al. 1999, Heckenberger et al. 2003]. O RAPD pode ser muito útil numa análise preliminar da variabilidade genética, e também na identificação de proveniências [Werner et al. 1997, Bordács & Burg 1997], mas nem sempre na distinção entre espécies e seus híbridos [González-Rodríguez et al. 2004a]; uma vez sequenciados os loci RAPD de maior utilidade, podem adoptar-se primers selectivos de modo a evidenciá-los, melhor e com maior reprodutibilidade, para análises genéticas [Bodénès et al. 1997]. Uma estratégia alternativa de obter sequências selectivas é a análise de diferença representacional [Zoldos et al. 2001]. Já os AFLPs, atraentes pela elevada quantidade de informação que trazem numa só electroforese para cada indivíduo, parecem ser mais eficazes para a variação intra-específica do que na diferenciação entre espécies próximas, pelo menos nos Quercus [Cervera et al. 2000, Kashani & Dodd 2002, Mariette et al. 2002, Kelleher et al. 2005]; além disso, se o enzima de restrição usado na segunda digestão no protocolo AFLP for sensível à metilação (donde, supõe-se, à regulação da expressão dos genes) podem tornar-se mais evidentes as diferenças entre genomas de espécies próximas [Scotti-Saintagne et al. 2004]. Pela cobertura intensa que fazem do genoma, os marcadores de RAPD como os dos AFLPs estão sujeitos a efeitos de selecção direccional por desequilíbrio de ligação com loci adaptativos (efeito hitch-hike [Mariette et al. 2002, Volis et al. 2003]). Por outro lado, é notável a sua utilização como marcadores da resistência poligénica a um patogénio exótico [Dodd et al. 2005].
Cabe ainda referir os marcadores ISSR (inter simple sequence repeats), que seguem um princípio análogo aos marcadores de RAPD, excepto que os primers, semi-arbitrários, hibridam com sequências simples repetidas (micro-satélites) [Zietkiewicz et al. 1994]; esta técnica permitiu, por exemplo, aperfeiçoar a interpretação de híbridos no género Penstemon [Wolfe et al. 1998].
Os SSCP (single-strand conformation polymorphisms) e os loci SSR (simple sequence repeats, também chamados “micro-satélites”) são marcadores em geral não associados a segmentos funcionais do genoma, altamente polimórficos e codominantes, permitindo elevadas probabilidades de exclusão, mesmo descontando o chamado “fluxo genético críptico”, suficientes para determinar os progenitores de cada indivíduo, entre outras determinações [Bodénès et al. 1997, Dow & Ashley 1997, Isagi 1997, Lexer et al. 1999, Aldrich et al. 2003, Cottrell et al. 2003, Muir & Schlötterer 2005]. Ambos os tipos de marcadores exigem um grande esforço inicial de rastreio do genoma, especialmente se se pretende fazer uma cobertura intensiva do genoma [Heckenberger et al. 2003], mas uma vez estabelecidos, o facto de dependerem de primers específicos para sequências em geral bem conservadas permite uma “transferência” para espécies congenéricas não testadas que tem sido, pelo menos nos loci SSR nucleares, muito bem-sucedida [Hornero et al. 2001]. Contudo, isto não exclui a necessidade de aumentar o número de loci SSR, que é desejável a todos os títulos [Muir & Schlötterer 2005]. Os alelos nos loci SSR distinguem-se pelo número de repetições da sequência “simples” que contêm, e a taxa de mutação por duplicação ou deleção é invulgarmente elevada [Chakraborty et al. 1997]; em princípio aplica-se o modelo de mutação stepwise, com estatísticas próprias, seja adaptando os conceitos de distância genética [Goldstein et al. 1995, Shriver et al. 1995] ou do FST em termos de tempo de coalescência de linhagens evolutivas [Slatkin 1995], mas a comparação entre estatísticas no tratamento dos dados nem sempre confirma as vantagens desse modelo [Goldstein et al. 1995, Cottrell et al. 2003]. A possibilidade de haver um marcador SSR com um polimorfismo demasiado elevado em relação aos restantes pode originar distorções na análise [Walter & Epperson 2004]. Pode economizar-se na utilização de marcadores SSR juntando vários loci na mesma análise electroforética [Hornero et al. 2001, Dzialuk et al. 2005].
O elevado grau de polimorfismo SSCP deve-se à multiplicidade de variações por substituição pontual ou por inserção/deleção que as sequências (por sinal relativamente curtas) comportam [Nataraj et al. 1999]. Se de espécies diferentes, moléculas comigrantes num gel SSCP são provavelmente homoplásicas, e não o mesmo “alelo” como por vezes se supõe [Bodénès et al. 1997], o que tem de verificar-se ao nível da sequência nucleotídica. Utilizando um princípio semelhante à análise de SSCPs, a análise de heteroduplexos terá até algumas vantagens [Nataraj et al. 1999], mas tanto uma técnica como outra têm permanecido em segundo plano, apesar da sua relativa simplicidade operacional.
Quanto ao DNA citoplásmico, mitocondrial ou plastidial, tem três particularidades muito convenientes, que são a existência de múltiplas cópias por célula, a provável neutralidade selectiva da variação, e a hereditariedade uniparental (sem recombinação, portanto). Esta última confere um Ne proporcionalmente inferior ao dos genes nucleares em 4 vezes, e consequentemente uma maior taxa de fixação de que resultam estatísticas de diferenciação populacional muito mais elevadas; foi através desta abordagem que se abriram novas possibilidades para uma reconstrução das rotas migratórias à escala de milhares de anos [Hewitt 1999], ou se detectaram expansões mediadas pela acção humana [Fineschi et al. 2000, Cottrell et al. 2003, Lumaret et al. 2005]. O facto de poder considerar-se em bloco toda a informação do DNA citoplásmico, isto é, como um haplótipo (ou “alelos ordenados”), torna-o particularmente adequado à aplicação da estatística NST, na qual as distâncias genéticas entre haplótipos contribuem para a estimativa da diferenciação genética intrapopulacional (o GST é o caso particular em que as distâncias entre haplótipos são unitárias) [Pons & Petit 1996]. Paradoxalmente, é na distinção entre espécies que este tipo de marcadores não tem grande utilidade, senão a de evidenciar uma extensa partilha de caracteres, como foi discutido anteriormente para os Quercus (parte II, secção D2, e parte III, secção C2 [Whittemore & Schaal 1991, Petit et al. 1997, Dumolin-Lapègue et al. 1997a, Bordács 2000, Belahbib et al. 2001, Collada et al. 2002, Finkeldey & Mátyás 2003, González-Rodríguez et al. 2004b, Lumaret et al. 2005]), e também se constata noutros grupos [Clark et al. 2000, Vendramin et al. 2000].
Sendo todos estes marcadores amostragens dos polimorfismos ao nível de sequência nucleotídica, o estudo directo destes (single nucleotide polymorphisms ou SNPs, pequenas inserções e deleções), tornado relativamente acessível com os desenvolvimentos no estudo de genomas, deverá constituir o padrão de referência para todos os outros marcadores. No contexto da inferência filogenética, os loci de rDNA têm um já longo historial e também foram estudados nos Quercus [Manos et al. 1999, 2001, Bellarosa et al. 2005], porém a análise das sequências requer algumas precauções [Mayol & Rosselló 2001, Bellarosa et al. 2005].
Dentro deste panorama, são vários os problemas associados à utilização dos polimorfismos enzimáticos. Além da grave questão que constitui a interpretação ecológica dos polimorfismos, que só no caso de serem neutrais podem ser extrapoláveis para o restante genoma onde também se aplique esse pressuposto, há a questão da amostragem que fazem do genoma [DeWoody & DeWoody 2005]. Assim, estão em causa apenas alguns genes que codificam proteínas evidenciáveis num gel após electroforese, na sua maior parte enzimas suficientemente abundantes para os quais existam métodos de revelação [Herzog, 1996]. A esta selectividade da amostragem acresce a reduzida sensibilidade desta metodologia, dependente de substituições de resíduos de aminoácidos com alteração da carga eléctrica. Embora seja corrente considerar-se que essa redução atinge ¾ do total [Chakraborty & Nei 1976, Nei 1987], a partir das matrizes de substituição BLOSUM [Henikoff & Henikoff 1992] deverá considerar-se mais correcto um valor à volta de 60% (cf. Apêndice II secção B para os cálculos), isto é, em cada 5 substituições há em média 3 que não implicam, nas posições em que ocorrem, uma mudança de carga eléctrica. A escolha dum ou outro valor, dentro da perspectiva da variação neutral, afecta sobretudo a estimativa da taxa de substituição dos alelos nas populações e consequente calibração do chamado relógio molecular [Nei, 1987]. Já em géis de poliacrilamida a geometria dos isoenzimas também contribui para separá-los, podendo detectar-se mais variações a nível polipeptídico, possivelmente até ao dobro [Johnson 1979], o mesmo se passando com a focagem isoeléctrica. Em todos os casos, será difícil considerar que algum estudo dos fundos genéticos das populações através de polimorfismos em loci enzimáticos constitua uma amostragem satisfatória do real grau de polimorfismo existente. Ao que parece, mesmo os marcadores SSR e até os SNPs são limitados [DeWoody & DeWoody 2005].
No lado positivo, a universalidade dos métodos de revelação para cada actividade enzimática faz com que os loci enzimáticos possam ser estudados em qualquer espécie praticamente sem requererem um trabalho preliminar de detecção e validação de marcadores genéticos, que as abordagens baseadas no DNA impõem em maior ou menor grau. Há a questão de manter as actividades enzimáticas em solução até ao passo da revelação, que nas plantas pode ser bastante problemática como discutido anteriormente; mas uma vez ultrapassado esse obstáculo, o atractivo de ter potencialmente à disposição dezenas de loci para cada amostra é de grande relevância, a que se juntam a sua já longa utilização e a economia de implementação em amostragens de grandes dimensões [May 1991, Chavarriaga-Aguirre et al. 1999, Sork et al. 1999].
2) Vantagens e limites para o problema científico a abordar
Para detectar híbridos de sobreiro com azinheira entre as descendências, e estimar a taxa de hibridismo que se encontra nos materiais de propagação, a abordagem a adoptar deverá maximizar a satisfação de critérios de eficiência definidos a priori, e que são:
Capacidade de discriminação entre sobreiro, azinheira e os respectivos híbridos. Idealmente, buscam-se loci monomórficos em cada uma das espécies, com genótipos diferentes (facilmente diferenciáveis) entre uma e outra, e com distinção dos genótipos heterozigóticos dos híbridos. Se a diferença dos fenótipos moleculares for muito pequena entre as duas espécies, podem surgir dificuldades de interpretação, especialmente na distinção dos genótipos híbridos. Loci polimórficos, numa ou nas duas espécies, podem ter uma variabilidade tal que haverá sempre um risco de certos fenótipos moleculares se sobreporem entre as duas espécies ou entre estas e os híbridos.
Os 2 critérios práticos para avaliar a capacidade discriminante dum locus são, como em qualquer procedimento de classificação de amostras, a especificidade e a sensibilidade, ou seja, as medidas de susceptibilidade aos falsos positivos e falsos negativos (respectivamente).
Uma amostra classificada como híbrido em função dum marcador genético, mas não o sendo, é um falso positivo. Um locus que produz falsos positivos perde especificidade como marcador genético. A especificidade a 100% (ausência de falsos positivos) é desejável, porém isso não obsta ao aproveitamento da informação dum locus se esta se julgar suficientemente específica, porque pode clarificar a informação fornecida por outros marcadores. No entanto, é necessária uma grande amostragem de populações-padrão, tanto maior quanto maior o número de alelos nesse locus em cada uma das espécies, para fazer esse julgamento.
Os falsos negativos são amostras cujos marcadores genéticos não evidenciam o seu hibridismo, apesar deste ser real. Um locus que produz falsos negativos perde sensibilidade como marcador genético. No caso da primeira geração de híbridos interspecíficos (F1), quanto maior o número de loci discriminantes utilizados, mesmo que individualmente não sejam 100% sensíveis, menor é o risco de ocorrerem falsos negativos. Contudo, à medida que são mais remotas das F1 que lhes deram origem, as gerações descendentes são cada vez mais difíceis de distinguir das espécies progenitoras da F1, por dois motivos: a progressiva diminuição da probabilidade de heterozigose interespecífica em cada locus discriminante, considerando que o padrão de reprodução é predominantemente o retrocruzamento com apenas uma das espécies progenitoras da F1; e a presumível selecção disruptiva nas linhagens descendentes dos F1 [Rieseberg & Linder 1999, Nason et al. 1992], que reduz ainda mais a dita probabilidade. Na realidade, apenas a análise de muitos loci (se possível, distribuídos uniformemente por todo o genoma), e preferencialmente por complementaridade entre diferentes métodos de análise, pode permitir uma razoável sensibilidade em gerações remotas.
Acresce que, na prática, a abordagem adoptada deve garantir uma percentagem de indeterminação tão baixa quanto possível, e a máxima reprodutibilidade, para que a especificidade e a sensibilidade não sejam comprometidas.
Custo de aplicação em larga escala. No rastreio de populações ou materiais de propagação florestais, as respostas esperadas dependem da capacidade de produzir informação para um grande número de amostras, que podem ser na ordem de vários milhares. Os custos a ponderar são de dois tipos: consumíveis (reagentes, etc.) e equipamentos, por um lado, e tempo de execução por outro.
Investimento experimental prévio. A cada método correspondem requisitos diferentes de aplicação, seja na sua implementação inicial (dificuldades intrínsecas ao método) seja na sua adaptação ao problema particular, neste caso o da detecção de hibridismo. A generalidade dos marcadores de DNA úteis são fruto duma selecção prévia que pode ser bastante morosa, mas uma vez conseguida, se as sequências específicas utilizadas estiverem conservadas evolutivamente, pode adaptar-se prontamente a outras espécies congenéricas.
Dentro deste conjunto de critérios, as diferentes classes de marcadores de DNA em sobreiro e azinheira poderão oferecer vantagens como sofrer limitações. No caso do DNA citoplásmico, a ausência de discriminação entre espécies, repetidamente evidenciada dentro do género Quercus (cf. Parte II secção D2), não pode mais do que apontar o sentido predominante da hibridação à escala geológica, sem com tal permitir qualquer estimativa do grau de introgressão, pois a uniparentalidade da hereditariedade citoplásmica não se correlaciona com a transmissão dos genes nucleares, tal que tanto pode haver praticamente “puros” no genoma nuclear mas com DNA citoplásmico da outra espécie, assim como os pode haver com DNA citoplásmico da própria espécie mas com forte introgressão do genoma nuclear da outra espécie.
Marcadores SSR ou SSCP têm a desvantagem, neste contexto, de serem muito polimórficos. Por isso a “transferência” de marcadores SSR, de Q. myrsinifolia Blume (= Q. glauca Thunb.), Q. macrocarpa, Q. petræa e Q. robur, para sobreiro [Hornero et al. 2001], e sobreiro e azinheira [Soto et al. 2003], que por certo terá grandes aplicações onde este tipo de marcadores têm revelado maior utilidade (análise de parentesco, medição directa do fluxo genético), não deverá ter grande potencial para o problema aqui em análise. Se se comprovar que o elevado polimorfismo nos loci SSR flutua à volta de valores mais ou menos constantes entre espécies congenéricas [Hornero et al. 2001], então será fácil encontrarem-se em cada locus alelos que numa das espécies têm migração mais rápida que os da outra, e outros de migração mais lenta, e nesses loci a discriminação, embora teoricamente possível, pode ficar dificultada pelo requisito de exactidão na estimativa do tamanho dos segmentos, nomeadamente pela uniformidade da calibração das electroforeses (o mesmo problema pode surgir com os marcadores SSCP, mas talvez não tão sistematicamente). Além disso, as “bandas-fantasma”, comuns em marcadores SSR, praticamente invalidam diferenças de 1 ou mesmo 2 pares nucleotídicos (bp) [Maehara et al. 2001]. Assim, dos 6 loci SSR nucleares propostos como discriminantes entre sobreiro e azinheira [Soto et al. 2003], apenas um (MSQ13) parece ter um bom potencial para este fim (ainda assim, o facto de se tratar duma amostragem de apenas 18 genes de sobreiro e 22 de azinheira deixa em aberto a possibilidade de serem encontrados novos alelos em cada locus até aí considerado discriminante, cf. parte III secção D3).
Marcadores RAPD ou AFLP, onde a regra de dominância completa é quase universal para os respectivos loci (isto é, os heterozigóticos não se distinguem dum dos homozigóticos), só podem ser úteis se o alelo dominante for nalguns casos o de sobreiro, noutros de azinheira, pois deste modo o hibridismo evidencia-se por um padrão “cumulativo” entre os das duas espécies. Caso esta “alternância da dominância” se verifique, o tipo de amostragem feito do genoma pelos AFLPs e a reprodutibilidade do método permitirão esperar um potencial de detecção de vestígios de hibridismo que nenhum outro método aqui tratado deverá mostrar [Dodd et al. 2002]. A possibilidade de introduzir pequenos melhoramentos no desenho dos primers [Cervera et al. 2000] ainda permite auspiciar para os marcadores AFLP maiores vantagens para a detecção de introgressão.
Comparativamente, os marcadores enzimáticos, mantendo as vantagens do ponto de vista económico e também a de pequeno investimento inicial (se não se contar com a questão genérica de conservar as actividades até ao momento da revelação), não têm sido em geral úteis para detectar hibridismos entre espécies próximas, por não serem suficientemente específicos [Rushton 1993, Kremer & Petit 1993, Zanetto et al. 1994, Tomlinson et al. 2000]. Mesmo assim, foi esta a abordagem proposta para os de sobreiro e azinheira com o projecto PAMAF 8153, que esteve na origem do presente trabalho; em parte, por não existir nenhum trabalho prévio com outro tipo de marcadores, mas também porque se assumia um maior grau de afastamento filogenético entre estas duas espécies, influenciado pelo esquema da Flora Europæa [Tutin 1964], o que daria a priori algumas esperanças para a existência de marcadores específicos analisáveis com esta técnica.